最小生成树算法(Prim+Kruskal)
贪心算法四个字总结:目前最优图的一些概念具体看先前的一篇文章https://www.wztlink1013.com/blog/gqpli5/连通图在图论中,连通图基于连通的概念。在一个无向图 G 中,若从顶点i到顶点j有路径相连(当然从j到i也一定有路径),则称i和j是连通的。如果 G 是有向...
贪心算法四个字总结:目前最优
图的一些概念
具体看先前的一篇文章https://www.wztlink1013.com/blog/gqpli5/
连通图
在图论中,连通图基于连通的概念。在一个无向图 G 中,若从顶点i到顶点j有路径相连(当然从j到i也一定有路径),则称i和j是连通的。如果 G 是有向图,那么连接i和j的路径中所有的边都必须同向。如果图中任意两点都是连通的,那么图被称作连通图。如果此图是有向图,则称为强连通图(注意:需要双向都有路径)。图的连通性是图的基本性质。
生成树
包含图的全部顶点,边数最少的连通子图
最小生成树
总权值最小的生成树
常见问题(该算法)就是求最小生成树。
并查集
是一个数据结构,功能有查找a和b是否为同一组;将a和b合并为同一组。
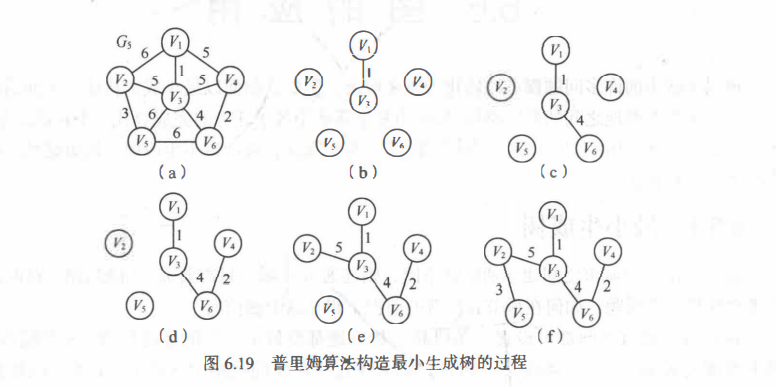
Prim算法思路
Prim——普里姆算法
类似于图的深度优先遍历一样,在遍历到一个结点的时候,在此根据该节点所连通的各边权值,取最小的,以此往复
Kruskal算法思路
Kruskal——克鲁斯卡尔算法
把所有边按照权值全部按数值大小拿出来,然后按顺序选取每条边,利用并查集的思想,如果这条边的两个端点不属于同一集合,那么就将它们合并,直到所有的点都属于同一个集合为止。
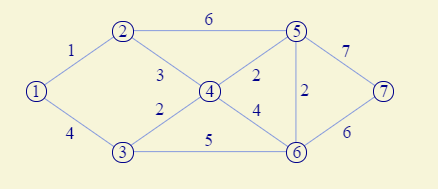
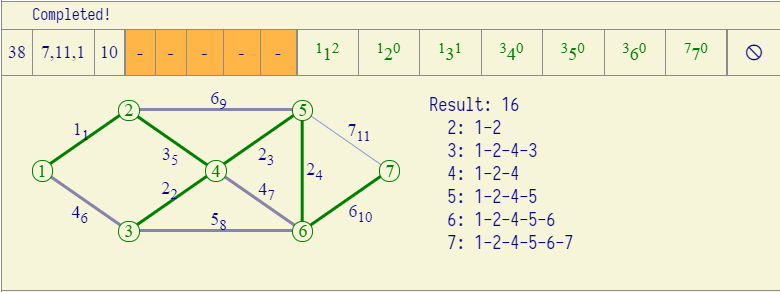
比如有如下这么一个图:

单独分析①②边和③④边情况下,两个不在一个集合里面,
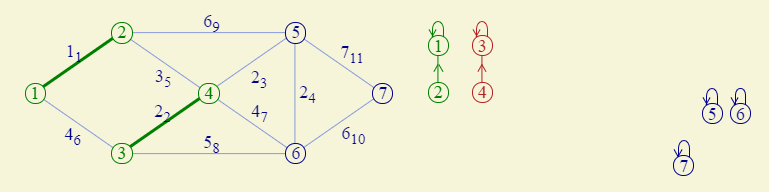
不断重复,不断判断是否为同一个集合,不在同一个集合的话,就合并,持续如此。比方说当一直操作到权值为3的时候,此时就需要将左右两个集合合并了
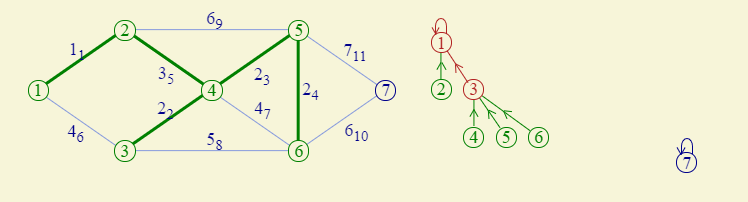
最后的结果样式就为如下
代码实现
Kruskal算法代码
#include <stdio.h>
#include <vector>
#include <algorithm>
namespace NS_KruskalMST {
using namespace std;
void KruskalMST();
int FindSet(int u);
void UnionSets(int u, int v);
void Initialization();
void GenEdges();
void MakeSets();
void Output(int v0);
#define INF INT_MAX
static int n;
static vector<vector<int>> WMatrix;
static vector<pair<pair<int, int>, int>> Edges;
//Node struct for the disjoint set
struct DJSNode {
int Parent; int Rank;
DJSNode(int p) : Parent(p), Rank(0) {}
};
static vector<DJSNode> DisjointSet;
static vector<pair<int, int>> MST;
//The adjacency list for MST
static vector<vector<int>> MSTList;
static vector<int> Prev;
void KruskalMSTCaller(int an,
vector<vector<int>> &wMatrix, int v0)
{
n = an;
WMatrix = wMatrix;
Initialization();
KruskalMST();
Output(v0);
}
void KruskalMST()
{
for (auto &e: Edges)
{
int u = e.first.first;
int v = e.first.second;
int setU = FindSet(u);
int setV = FindSet(v);
if (setU != setV)
{
MST.push_back(e.first);
if (MST.size() == n - 1)
break;
UnionSets(setU, setV);
}
}
}
int FindSet(int u)
{
while (u != DisjointSet[u].Parent)
u = DisjointSet[u].Parent;
//For path compression:
//DisjointSet[u].Parent =
// FindSet(DisjointSet[u].Parent);
return u;
}
void UnionSets(int u, int v)
{
if (DisjointSet[u].Rank >= DisjointSet[v].Rank)
DisjointSet[v].Parent = u;
else
DisjointSet[u].Parent = v;
if (DisjointSet[u].Rank == DisjointSet[v].Rank)
DisjointSet[u].Rank++;
}
void Initialization()
{
GenEdges();
sort(Edges.begin(), Edges.end(),
[](pair<pair<int, int>, int>a,
pair<pair<int, int>, int>b)
{return a.second < b.second; });
MakeSets();
MST.clear();
}
void GenEdges()
{
Edges.clear();
//Traverse the upper triangle of WMatrix
for (int i = 0; i < n - 1; i++)
{
for (int j = i + 1; j < n; j++)
if (WMatrix[i][j] != INF)
Edges.push_back({ {i, j},
WMatrix[i][j] });
}
}
void MakeSets()
{
DisjointSet.clear();
for (int i = 0; i < n; i++)
DisjointSet.push_back(DJSNode(i));
}
void OutputWMatrix()
{
printf("n = %d\n", n);
printf("The weight matrix:\n");
printf("%3c", ' ');
for (int j = 0; j < n; j++)
printf("%3d", j + 1);
printf("\n");
for (int i = 0; i < n; i++)
{
printf("%3d", i + 1);
for (auto j : WMatrix[i])
if (j < INF)
printf("%3d", j);
else
printf("%3c", '*');
printf("\n");
}
}
void OutputPath(int u)
{
if (Prev[u] == -1)
printf("%d", u + 1);
else
{
OutputPath(Prev[u]);
printf("-%d", u + 1);
}
}
void GenMSTList()
{
MSTList.clear();
MSTList.resize(n);
for (auto &e: MST)
{
MSTList[e.first].push_back(e.second);
MSTList[e.second].push_back(e.first);
}
}
void GenPrev(int v)
{
for (auto &u : MSTList[v])
if (u != -1)
{
Prev[u] = v;
auto w = find(MSTList[u].begin(),
MSTList[u].end(), v);
MSTList[u][w - MSTList[u].begin()] = -1;
GenPrev(u);
}
}
void Output(int v0)
{
printf("Kruskal's MST algorithm\n");
OutputWMatrix();
int wSum = 0;
for (int i = 0; i < n - 1; i++)
wSum += WMatrix[MST[i].first][MST[i].second];
GenMSTList();
Prev.clear();
Prev.resize(n);
Prev[v0] = -1;
GenPrev(v0);
printf("The MST edges:\n");
printf("Edge Weight\n");
for (auto &e : MST)
printf(" %d-%d %d\n", e.first + 1, e.second + 1,
WMatrix[e.first][e.second]);
printf("Total MST weight: %d\n", wSum);
printf("The MST paths from vertex %d:\n", v0 + 1);
for (int u = 0; u < n; u++)
if (u != v0)
{
printf("%3d: ", u + 1);
OutputPath(u);
printf("\n");
}
printf("\n");
}
} //namespace NS_KruskalMST
using namespace NS_KruskalMST;
void TestKruskalMST(int v0 = 0)
{
vector<vector<vector<int>>> w = {
//https://www.geeksforgeeks.org/
//prims-minimum-spanning-tree-mst-greedy-algo-5/
{
{ 0, 2,INF, 6,INF },
{ 2, 0, 3, 8, 5 },
{ INF, 3, 0,INF, 7 },
{ 6, 8,INF, 0, 9 },
{ INF, 5, 7, 9, 0 }
},
// Dijkstra's algorithm on Wikipedia
{
{ 0, 7, 9,INF,INF, 14 },
{ 7, 0, 10, 15,INF,INF },
{ 9, 10, 0, 11,INF, 2 },
{ INF, 15, 11, 0, 6,INF },
{ INF,INF,INF, 6, 0, 9 },
{ 14,INF, 2,INF, 9, 0 },
},
//https://www.geeksforgeeks.org/
//kruskals-minimum-spanning-tree-using-stl-in-c/
{
{ 0, 4,INF,INF,INF,INF,INF, 8,INF },
{ 4, 0, 8,INF,INF,INF,INF, 11,INF },
{ INF, 8, 0, 7,INF, 4,INF,INF, 2 },
{ INF,INF, 7, 0, 9, 14,INF,INF,INF },
{ INF,INF,INF, 9, 0, 10,INF,INF,INF },
{ INF,INF, 4, 14, 10, 0, 2,INF,INF },
{ INF,INF,INF,INF,INF, 2, 0, 1, 6 },
{ 8, 11,INF,INF,INF,INF, 1, 0, 7 },
{ INF,INF, 2,INF,INF,INF, 6, 7, 0 },
},
};
int k = w.size();
for (int i = 0; i < k; i++)
{
if (v0 > w[i].size() - 1)
v0 = w[i].size() - 1;
KruskalMSTCaller(w[i].size(), w[i], v0);
}
}
文章评论区
欢迎留言交流